- Author: Mark Bolda
Bajo la dirección de USDA-ARS (el servicio de agricultura del gobierno Federal de los EEUU) y otros investigadores, además la ayuda de la Comission de Fresa de California y otros apoyadores, el laboratorio diagnóstico de la Extensión Cooperativa de Universidad de California en Salinas finalmente está utilizando métodos basada en ADN (la materia genética de los organismos vivos) para hacer diagnosis de enfermedades de plantas. Esta prueba se está implementando para detectar y confirmar enfermedades importantes en fresa y otros cultivos de la costa central. Este método puede cumplir el proceso diagnostico en pocas horas, en vez de lo usual de varios días dependiendo en propagar el patógeno en varios medios laboratorios para positivamente identificarlo.
La Necesidad
Una parte indispensable de la producción agrícola es el poder de hacer diagnosis de enfermedades de plantas en forma rápida, precisa y confiable. Sin saber cuál es el agente responsable por tal enfermedad, productores, asesores de control de plagas y otra gente del campo son obstaculizados en hacer decisiones propicias sobre el manejo de enfermedades. Enfermedades de los suelos se dificultan en identificar, ya que podriciones de las raíces, podriciones de las coronas, recaídas de planta y marchiteces vasculares se semejan mucho.
El Método
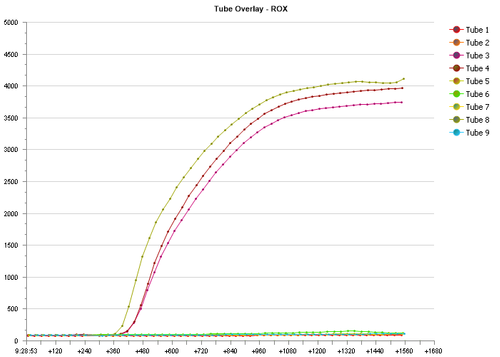
El método nuevo se conoce por sus siglas en inglés como “RPA” lo que quiere decir “amplificación de polimerasa recombinase”. El método RPA tiene componentes de ADN, lo que se llaman primer y sonda, que específicamente reconocen el ADN correspondiente en el patógeno bajo evaluación. Lo que deja el RPA perfecto para usar en un laboratorio de extensión agrícola, tal como tenemos en Salinas, es que RPA evalúa extractos de plantas crudas y maceradas (véase las fotos incluidas abajo). Otros tipos de evaluaciones basadas en el uso de ADN requieren extraer el ADN purificado del patógeno y para cumplir esto necesita aparatos caros y procedimientos más elaborados.
Patógenos detectados
Actualmente el laboratorio de extensión cooperativa de la Universidad de California brinda evaluaciones diagnosticas de RPA para dos patógenos. Phytophthora causa podriciones de raíces y coronas en fresa, frambuesa, además de un número fijo de verduras y unas plantas del monte. Nuestras pruebas de Phytophthora son específicos al género – quiere decir muchas variedades del Phytophthora que infectan muchos tipos de plantas- o especifico a la especie para los dos patógenos, P. cactorum y P. fragariae, que mayormente atacan a la fresa (Tabla 1). Nuestra otra prueba de RPA detecta el tipo específico de Macrophomina phaseolina que causa podrición severa y colapso de fresa. Por ser una prueba diseñada para el patógeno de fresa y no para otros tipos de Macrophomina que infestan otros cultivos, se llama esta prueba específica al genotipo (Tabla 1).
Este verano anticipamos añadir pruebas para el patógeno de marchitez de Verticillium, lo infecta a muchos cultivos más y marchitez de Fusarium de fresa causado por Fusarium oxysporum f. sp. fragariae. Todas las pruebas analizan materia infectada de planta solamente. Sin embargo, nosotros esperamos que nuestros esfuerzos continuados adaptarán RPA o métodos aún más sofisticados para detectar estos patógenos en los suelos.
Tabla 1. Pruebas de RPA actualmente disponible en el laboratorio de la Extensión Cooperativa de la UC.
|
Categoria de la prueba |
Pathógeno |
Comentarios |
|
Específico al género |
Phytophthora |
Detecta este patógeno en toda manera de planta tal como fresa, frambuesa, pimentón, espárragos, jitomate, avocado, plantas ornamentales y árboles del monte. |
|
Específico a la especie |
Phytophthora cactorum |
Primeramente para fresa pero este patógeno puede ser encontrado en unas plantas ornamentales. |
|
Específico a la especie |
Phytophthora fragariae |
Para fresa |
|
Específico al genotipo |
Macrophomina phaseolina |
Para Macrophomina que infecta fresa. |
Reconocimientos.
Este avance en diagnósticos de enfermedades rápidos y precisos fue desarrollado y implementado por el siguiente equipo de investigación y extensión: Mark Bolda (UC Extension, Santa Cruz Co.), Alyssa Burkhardt (USDA-ARS, Salinas), Oleg Daugovish (UC Extension, Ventura Co.), Steven Koike (UC Extension, Monterey Co.), Frank Martin (USDA-ARS, Salinas), Stacy Mauzey (UC Extension, Monterey Co.), Tim Miles (CSU Monterey Bay), and Cayla Tsuchida (UC Extension, Monterey Co.). Trabajo fue apoyado por California Strawberry Commission, California Avocado Commission CSU Agricultural Research Institute (fondo 5219101A), y elUSDA-California Department of Food and Agriculture Specialty Crop Block Grant Program (fondos SCB12051 y SCB14052 otorgado a F. Martin).